从零入门 AI for Science(AI+药物) 笔记
使用平台
我的Notebook · 魔搭社区 https://modelscope.cn/my/mynotebook/preset .
魔搭高峰期打不开Task3又换回飞桨了 吧torch 架构换成了 飞桨的paddle
飞桨AI Studio星河社区-人工智能学习与实训社区
https://aistudio.baidu.com/projectdetail/8191835?contributionType=1
主要操作
运行实例,如果有时长尽量选择方式二(以下操作基于方式二的实例实现)
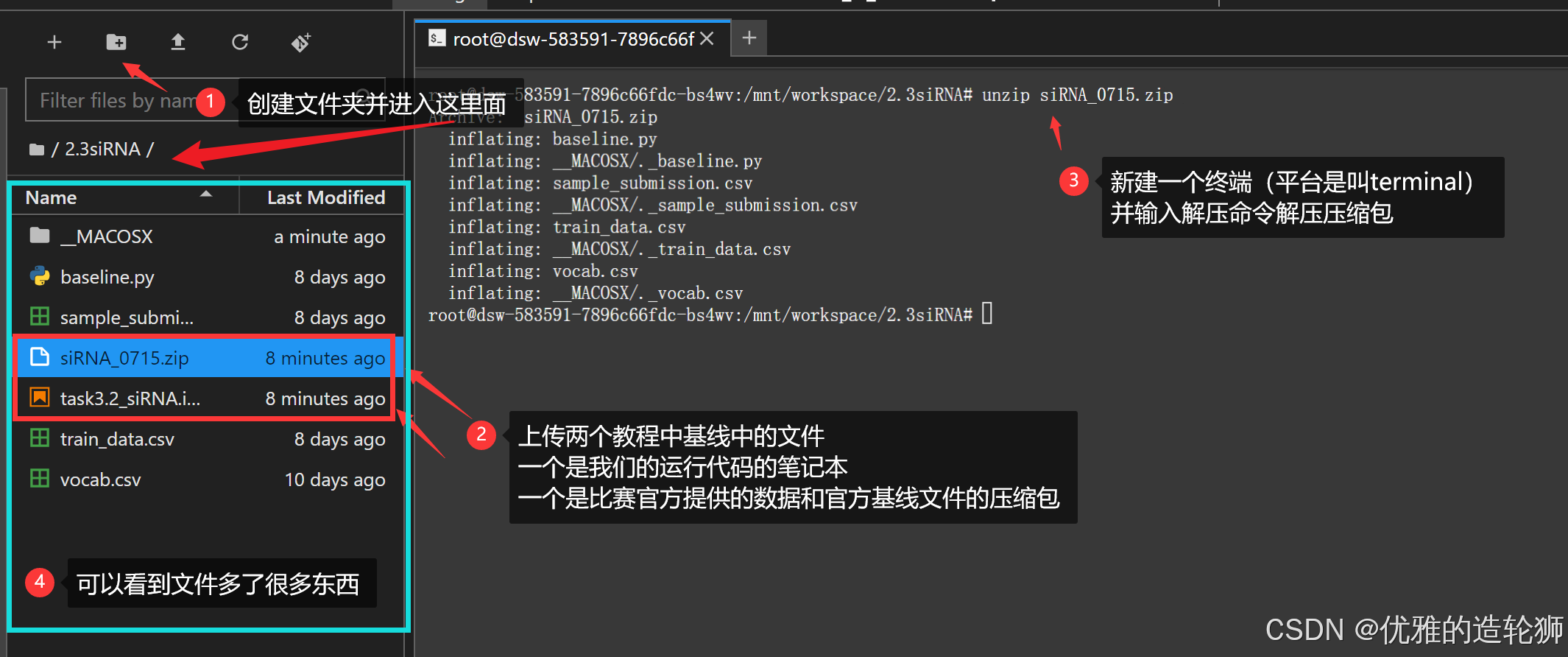
创建文件夹,并重命名为 2.3siRNA
上传两个文件
到文件夹, 这里面的第三个按钮是上传
在当前文件夹打开终端(如图示意打开终端)并输入解压命令
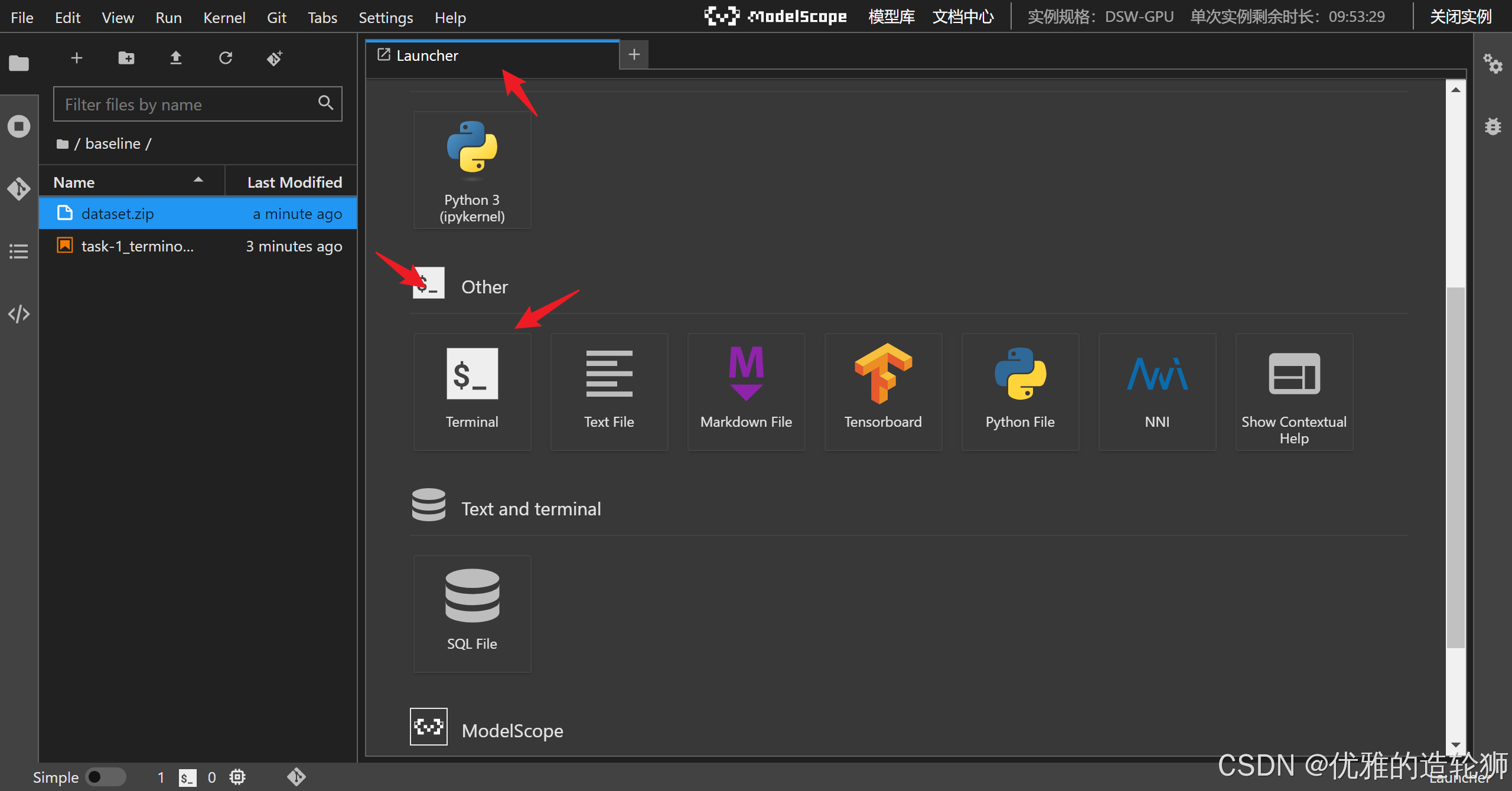
注意:如果你的压缩包名字不是这个请将“siRNA_0715.zip” 换成你的压缩文件的名字“xxx.zip”(xxx为文件名)
(方便复制)
1 | unzip siRNA_0715.zip |
到这里准备工作可以了,如果解压出问题了,可以重新上传一下,然后重复解压的操作
总览
参赛平台
Task 1 跑通基线
baseline
- 运行笔记本
2.3siRNA/task3.2_siRNA.ipynb
就是这个橙不溜秋的书签,双击运行 - 运行笔记本中的所有代码
- 等待结果出来
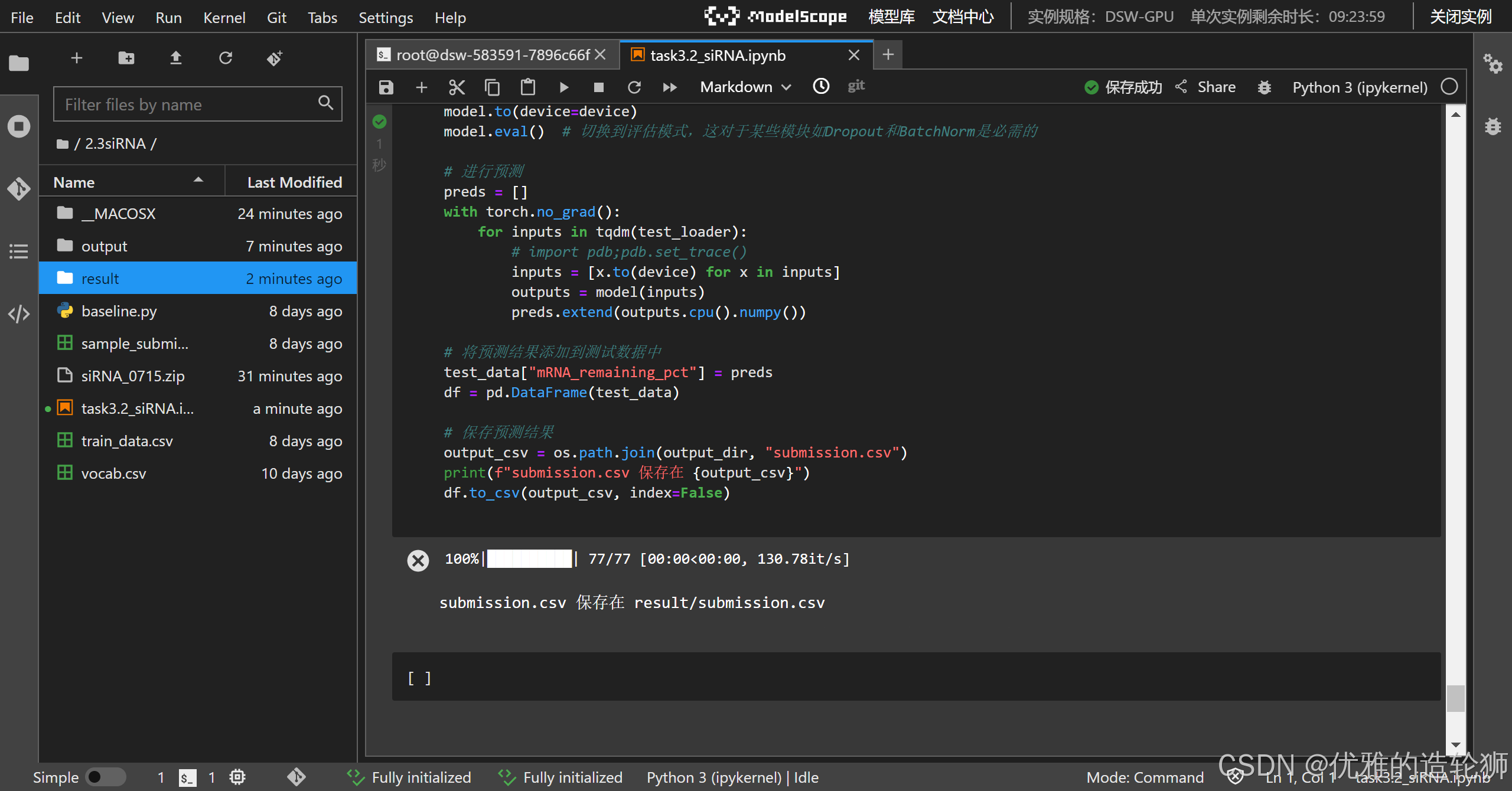
可以看到多了一个文件夹和文件右键下载result/submission.csv文件(download)
注意用完平台记得关闭实例(右上角)!!!
注意用完平台记得关闭实例(右上角)!!!
注意用完平台记得关闭实例(右上角)!!!

tips: 算力充足可以当我没说,不关的话时长会一直使用
提交文件获得第一个分数
平台: 上海科学智能研究院
注册和实名制略过
点击提交结果和选中刚刚下载的文件等待上传
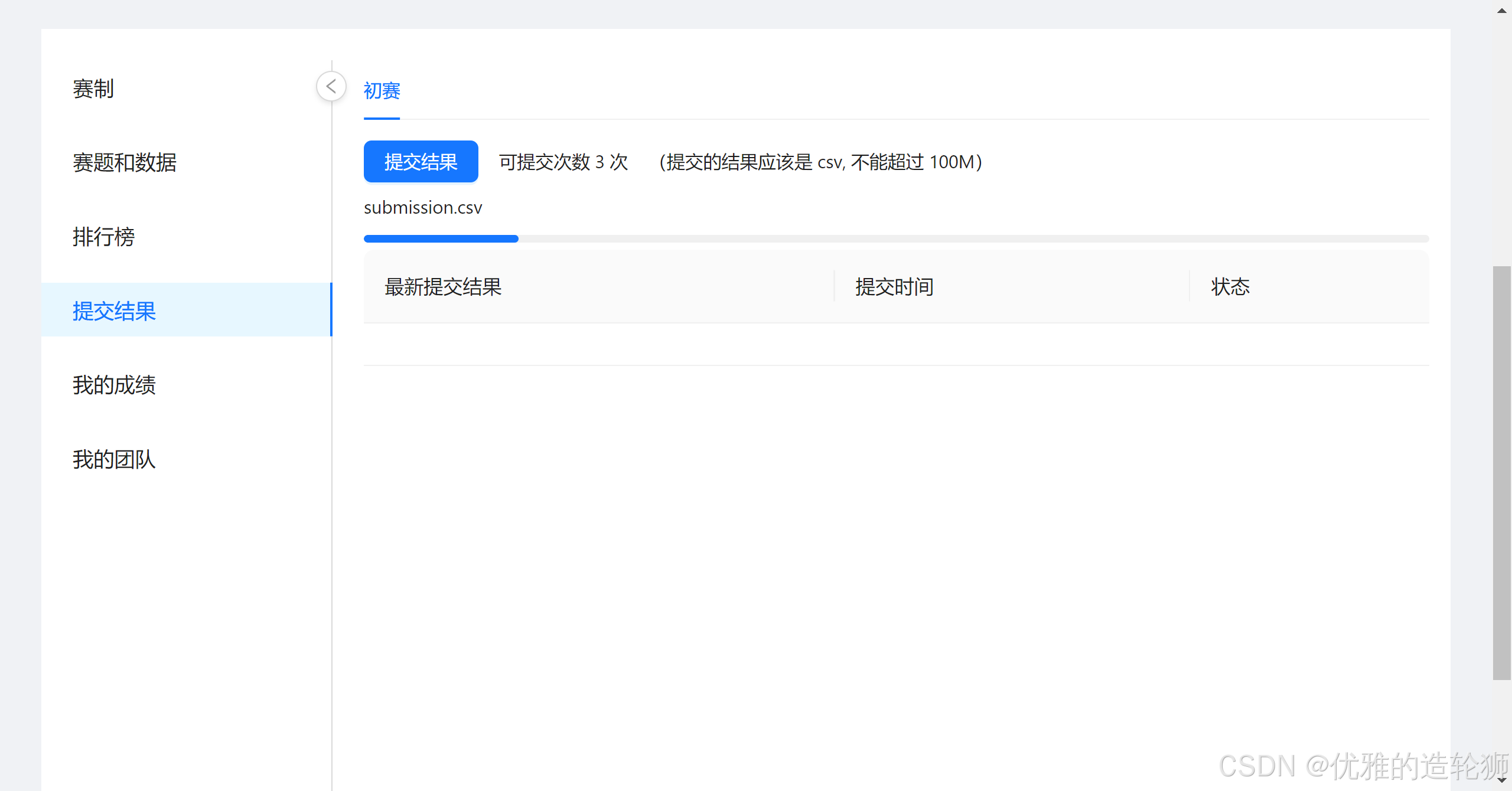
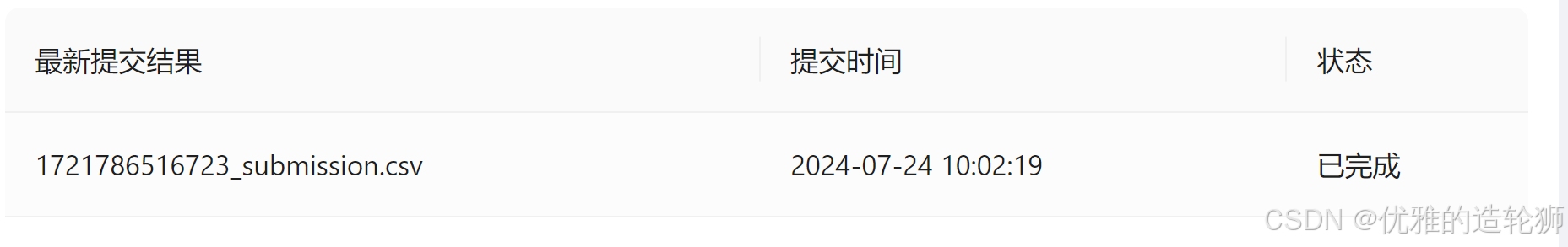
点击我的成绩查看分数
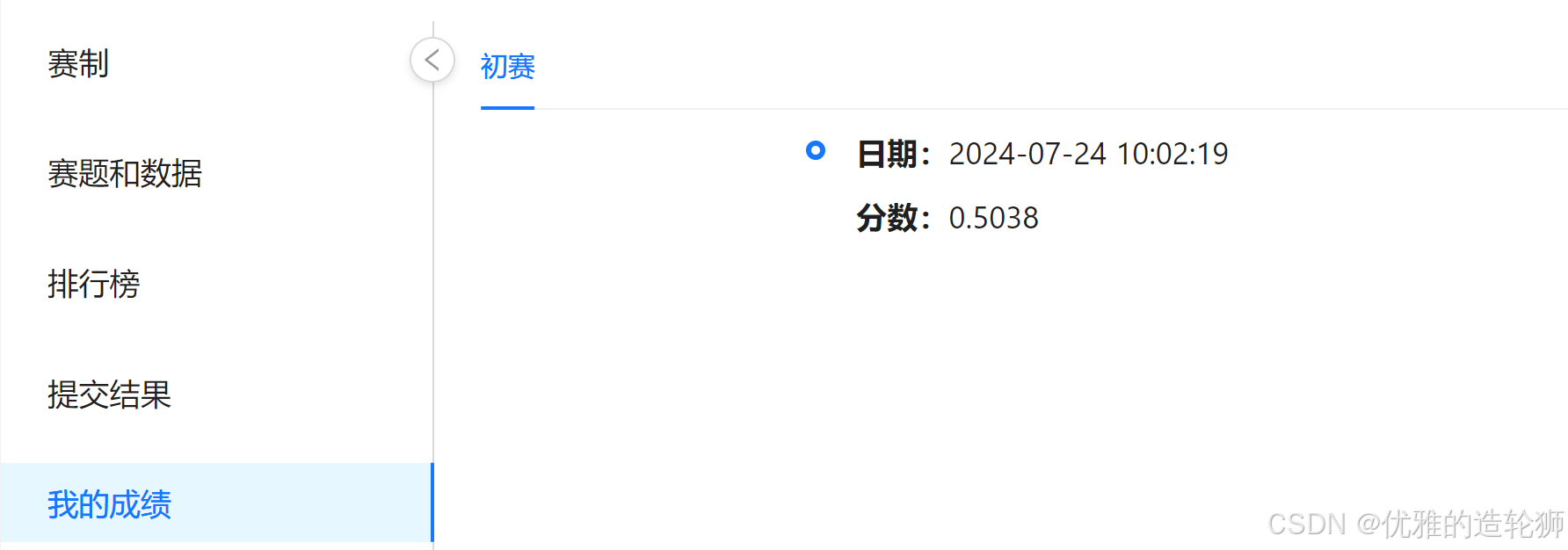
其中task1中只选择了部分作为特征值,可以将全部的有效数据转换成特征值,必涨点。
训练数据表头说明
数据来源于官网

特征的分析总结
| 特征类别 | 特征字段名称 | 特征描述 | 分析目的 |
|---|---|---|---|
| 基因特异性 | gene_target_symbol_name | 靶基因符号名称 | 研究不同基因名称对siRNA设计的影响 |
| gene_target_ncbi_id | 靶基因的NCBI标识 | 研究不同NCBI ID对siRNA设计的影响 | |
| gene_target_species | 靶基因参考序列的物种 | 研究不同物种对siRNA沉默效率的影响 | |
| siRNA序列特征 | siRNA_sense_seq | siRNA的sense序列 | 分析sense序列设计对沉默效率的影响 |
| siRNA_antisense_seq | siRNA的antisense序列 | 分析antisense序列设计对沉默效率的影响 | |
| modified_siRNA_sense_seq | 带修饰的siRNA的sense序列 | 分析修饰对siRNA功能的影响 | |
| modified_siRNA_antisense_seq | 带修饰的siRNA的antisense序列 | 分析修饰对siRNA功能的影响 | |
| siRNA浓度和单位 | siRNA_concentration | 实验使用的siRNA浓度 | 研究不同浓度对沉默效率的影响 |
| concentration_unit | siRNA浓度单位 | 研究不同单位对siRNA浓度影响的理解 | |
| 转染方法 | Transfection_method | 转染方法 | 分析不同转染技术对siRNA传递和沉默效果的影响 |
| 转染后持续时间 | Duration_after_transfection_h | 转染后持续时间 | 了解转染后不同时间点的沉默效果 |
| 序列分解列表 | modified_siRNA_sense_seq_list | 带修饰的siRNA的sense序列分解列表 | 识别关键核苷酸位点,优化siRNA设计 |
| modified_siRNA_antisense_seq_list | 带修饰的siRNA的antisense序列分解列表 | 识别关键核苷酸位点,优化siRNA设计 | |
| 靶基因序列 | gene_target_seq | 靶基因的参考序列 | 分析siRNA与靶基因序列匹配程度对沉默效率的影响 |
| 沉默效率 | mRNA_remaining_pct | 实验后mRNA的剩余百分比 | 评估不同条件下siRNA沉默效率的直接指标 |
目前尝试了 计算序列的长度 、计算序列的熵值、序列中腺嘌呤(A)、胸腺嘧啶(T)、胞嘧啶(C)和鸟嘌呤(G)的数目、GC含量、序列的熵值
创建了两个函数作为特征
calculate_sequence_features函数:- 它首先计算序列的长度。
- 然后计算序列中腺嘌呤(A)、胸腺嘧啶(T)、胞嘧啶(C)和鸟嘌呤(G)的数目,并由此计算出它们的相对频率。
- 接着计算GC含量,即序列中G和C的比例,这是影响DNA稳定性的一个重要因素。
- 计算序列的熵值,熵是一个度量序列随机性或复杂性的指标。熵越高,表示序列的多样性越高,没有明显的偏好性。
calculate_entropy函数:- 计算序列中每个核苷酸(A、C、G、T)的数目。
- 用一个字典来存储每个核苷酸的计数。
- 遍历这个字典,对每个非零计数的核苷酸,使用公式 $-p \log_2(p)$ 来计算其对熵的贡献。
(比赛原因先不贴代码)
至此Task1 baseline 任务完成
Task1 知识点终结
基因组分词器类
基因组分词器的目的是将基因组序列分割成固定长度的n-gram片段。这是为了进一步处理或分析基因组数据时的需要。
基因组数据通常是由ACGT四个字母(腺嘌呤、胞嘧啶、鸟嘌呤和胸腺嘧啶)组成的序列。
n-gram
指由n个连续字母构成的片段。将基因组序列分割成n-gram片段可以帮助我们理解基因组的结构和功能。
基因组分词器将基因组序列分割成固定长度的n-gram片段可以用于以下应用:
- 基因组注释:通过分析n-gram片段可以识别基因、启动子、转录因子结合位点等功能区域。
- 基因组比对:将n-gram片段与已知的基因组序列进行比对,可以找到相似的片段并识别基因的同源性。
- 基因组序列分类:通过分析n-gram片段可以将不同物种的基因组序列进行分类。
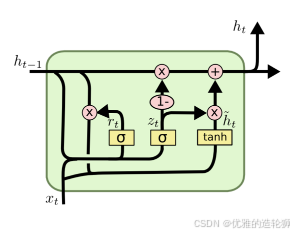
GRU的神经网络模型
GRU是一种循环神经网络(RNN)模型,全称为Gated Recurrent Unit。它是一种改进的RNN架构,用于处理序列数据,尤其在自然语言处理和语音识别等任务中表现出色。
GRU通过引入门控机制来解决传统RNN存在的短期记忆和长期记忆不平衡的问题。它具有两个门控单元:重置门(reset gate)和更新门(update gate)。重置门控制了当前状态如何与先前状态相结合,而更新门控制了用于传递信息的新状态的计算。
GRU单元结构如下图所示
GRU是Ilya Sutskever和Oriol Vinyals等人在2014年提出的一种改进的RNN单元,它旨在解决传统RNN在处理长序列时出现的梯度消失或梯度爆炸问题。
GRU的核心思想是引入两个门控机制:更新门(Update Gate)和重置门(Reset Gate)。这两个门控机制允许模型动态地决定在每个时间步上应该保留多少之前的信息,以及应该更新多少当前的信息。这使得GRU能够更好地捕捉长距离依赖关系。
GRU的数学模型
更新门(Update Gate)
更新门决定了在当前时间步应该保留多少之前的隐藏状态。更新门的公式如下:
$$
z_t = \sigma(W_z \cdot [h_{t-1}, x_t])
$$
其中,$z_t$ 是更新门的输出,$W_z$ 是更新门的权重矩阵,$\sigma$ 是sigmoid函数(不懂的后面有讲 sigmoid函数)。
重置门(Reset Gate)
重置门决定了在当前时间步应该忽略多少之前的隐藏状态。重置门的公式如下:
$$
r_t = \sigma(W_r \cdot [h_{t-1}, x_t])
$$
其中,$r_t$ 是重置门的输出,$W_r$ 是重置门的权重矩阵。
候选隐藏状态(Candidate Hidden State)
候选隐藏状态是当前时间步的新信息,其公式如下:
$$
\tilde{h}t = \tanh(W \cdot [r_t \odot h{t-1}, x_t])
$$
其中,$\tilde{h}_t$ 是候选隐藏状态,$W$ 是候选隐藏状态的权重矩阵,$\odot$ 表示Hadamard乘积(不懂的后面有讲 Hadamard乘积)。
最终隐藏状态(Final Hidden State)
最终隐藏状态结合了之前保留的信息和当前的新信息,其公式如下:
$$
h_t = (1 - z_t) \odot h_{t-1} + z_t \odot \tilde{h}_t
$$
其中,$h_t$ 是最终的隐藏状态。
GRU在自然语言处理、语音识别和时间序列预测等领域有着广泛的应用。
小结
相比于普通的RNN模型,GRU具有更好的记忆能力和更强的建模能力,同时减少了参数数量,使得训练过程更加高效。 由于其优秀的性能和实用性,GRU已经成为经典的循环神经网络模型之一,并被广泛应用于各种序列数据分析任务中。
学科知识
RNA干扰(RNAi)
RNA干扰(RNAi)是一种细胞内的基因调控机制,通过通过RNA分子的干扰来抑制特定基因的表达。
RNAi在细胞内通过两种途径实现:小干扰RNA(siRNA) 和微小RNA(miRNA)。
在RNAi中,基因表达的抑制通常发生在转录后水平。当特定基因的DNA序列转录成RNA时,RNA聚合酶将生成多个复制的RNA分子。这些RNA分子中的一部分可以通过Dicer酶切割成长度约为21-23个核苷酸的小片段,即siRNA或miRNA。这些小片段与蛋白质复合物形成RNA-诱导沉默复合物(RISC),并通过与靶标mRNA相互作用来抑制其翻译或引起其降解。
siRNA是通过外源性引入细胞的siRNA分子,通过与特定基因的mRNA相互作用来抑制其表达。
miRNA是内在于细胞的小RNA分子,能够识别并与多个基因的mRNA结合,从而调节多个基因的表达。
RNAi在生物学研究中被广泛应用。可以用于研究基因功能,筛选潜在药物靶点,开发基因治疗方法等。还有潜力成为治疗疾病的方法,包括癌症、病毒感染和遗传疾病等。
Dicer 酶
RNA 干扰(RNAi)过程中的一个关键酶。它是一种 RNase III 家族的内切酶,在 RNAi 过程中起着重要的作用。
Dicer 酶能够识别和切割双链 RNA(dsRNA)分子,将其切割成短的双链小干扰 RNA(siRNA)。
RNAi作用机制
文档内容里面的这个讲的很详细我啃臭cv一份
生物体内,RNAi首先将较长的双链RNA加工和切割成 siRNA,通常在每条链的3’末端带有2个核苷酸突出端。负责这种加工的酶是一种RNase III样酶,称为Dicer。形成后,siRNA与一种称为RNA诱导的沉默复合物(RNAinduced silencing complex, RISC)的多蛋白组分复合物结合。在RISC复合物中,siRNA链被分离,具有更稳定的5′末端的链通常被整合到活性RISC复合物中。然后,反义单链siRNA组分引导并排列在靶mRNA上,并通过催化RISC蛋白(Argonaute family(Ago2))的作用,mRNA被切割,即对应基因被沉默,表达蛋白能力削弱。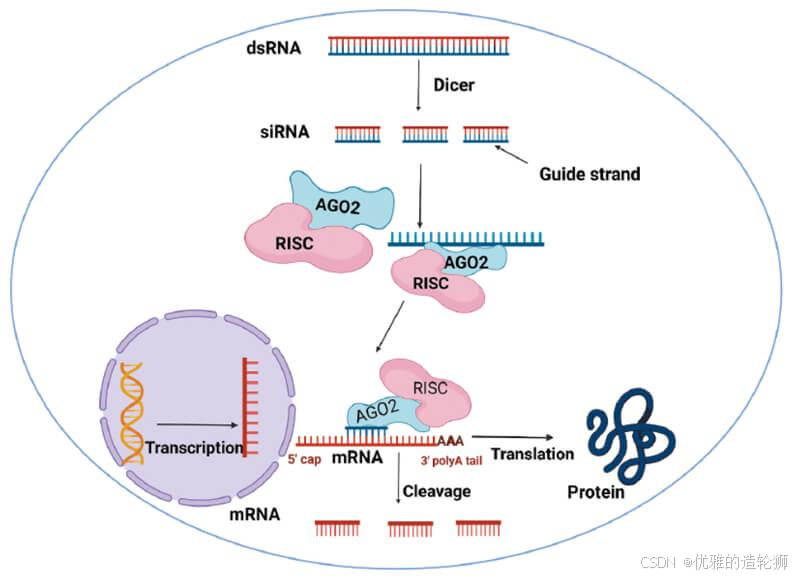
传统siRNA设计原则与知识
siRNA的沉默效率与众多因素相关,例如siRNA的稳定性、修饰、转染方法等。一些经验的生物知识可用于特征构建和AI模型的设计。
在siRNA一般设计过程中有以下知识和原则:
- siRNA序列(一般为反义链)与靶向RNA互补。
- siRNA序列长度一般在19~29nt之间。研究表明21nt相比27nt对靶基因的最大抑制率更容易达到。
- 一般来说,从靶基因起始密码子AUG下游50~100个核苷酸,或位于终止密码子50-100个核苷酸范围内的序列(确保转录基因为沉默状态)搜寻理想的siRNA序列,越靠近靶基因的3′端,其基因沉默效果可能越好。
- 一般设计好的潜在siRNA序列,会在GenBank数据库进行BLAST,去掉其他基因有显著同源性的靶序列(错误靶向)。
- 具体序列而言,最好为AA+(Nn)UU(N代表任意碱基,n为碱基数目),其次是NA(Nn)UU和NA(Nn)NN。
- 一般情况下,siRNA的稳定性直接影响其最终在细胞中的敲低效率。在siRNA的反义链5’端第一个碱基尽量可能是为A或U; siRNA正义链的5’端第一个碱基尽量为G或C。
- 一般情况下,3′端的2个碱基使用突出的dTdT(deoxythymidine dinucleotide)取代,能够增强siRNA 双链复合体的稳定性,进而增加siRNA的敲低效率。
- G/C含量在30%~52%的siRNA序列,其沉默基因效果较好。研究表明40–55% GC的含量敲低效率高于GC含量高于55%的。
- 一般来说,siRNA序列中连续2个及以上G/C能够降低双链RNA内在稳定性,从而降低siRNA在细胞中的敲低效率;而连续3个以上的A和U可能终止由RNA Polymerase III介导的转录作用。siRNA序列中的重复序列或回文结构可能形成发夹状结构,这种结构的存在可以降低siRNA敲低效率。
化学修饰siRNA
化学修饰siRNA是指通过在siRNA分子上引入化学修饰基团,改变其结构或性质的方法。这种修饰可以增强siRNA的稳定性、增加其目标特异性、改善细胞内进入能力等。
常用的siRNA化学修饰包括以下几种:
- 2’-氧甲基(2’-O-Me)修饰:这种修饰是将2’-羟基上的氧原子替换为甲基基团。它可以增加siRNA的稳定性,提高RNA酶的抵抗性。
- 2’-氟(2’-F)修饰:这种修饰是将2’-羟基上的氧原子替换为氟原子。它可以提高siRNA的稳定性和特异性,减少对非特定靶标的作用。
- 磷酸甲酯(PS)修饰:这种修饰是在磷酸二酯桥上引入甲酯基团。它可以增强siRNA的稳定性和细胞内进入能力。
- 枝状修饰:这种修饰是在siRNA分子上引入枝状结构,增加其稳定性和亲水性。
- 核苷酸修饰:这种修饰是在siRNA的碱基上引入修饰基团,例如甲基化、二硫苷化等。它可以改变siRNA与靶标RNA的配对能力和稳定性。
化学修饰siRNA可以优化其性能和提高其在RNAi研究和治疗中的应用潜力。但化学修饰可能会对siRNA的活性和毒性产生影响,因此在设计和选择修饰方案时需要进行全面的评估和优化。
机器学习知识点
MAE (Mean Absolute Error)
表示预测值与真实值之间的平均绝对误差。
它计算每个样本的预测值与真实值之间的差值的绝对值,然后对所有样本取平均。
召回率(Recall)
表示所有真正例中被正确预测为正例的比例。
召回率可以衡量模型对正例的覆盖程度,即模型有多少能够找到真正例。
F1得分
精确度和召回率的调和平均值。
F1得分的取值范围为0到1,其中1表示最佳性能,0表示最差性能。
精确度(Precision)
表示被预测为正例中实际为正例的比例。
精确度可以衡量模型的准确性,即模型有多少预测为正例的样本真正是正例。
赛题评分代码
1 | # score = 50% × (1−MAE/100) + 50% × F1 × (1−Range-MAE/100) |
综合评分
1 | score = (1 - mae / 100) * 0.5 + (1 - range_mae / 100) * f1 * 0.5 |
最终的评分是根据模型在三个方面的表现进行计算的。
- 通过计算平均绝对误差(MAE)来衡量模型的整体预测精度,MAE越小,表明模型的预测误差越小,得分越高。
- 通过计算在指定范围内的平均绝对误差(Range MAE),来衡量模型对于特定范围内的预测的准确性,Range MAE越小,表明模型在该范围内的预测误差越小,得分越高。
- 计算模型的分类性能,即精确度、召回率和F1得分。F1得分越高,表明模型在分类任务上的性能越好,得分越高。最终的评分是这几个值的加权平均数,其中MAE和Range MAE各占50%权重。
综合考虑这些因素,可以得出模型的总体表现得分。
小结
在分类问题中,精确度和召回率是互相影响的指标。高精确度可能意味着模型只预测那些非常确信的正例,导致召回率较低。相反,高召回率可能意味着模型会将更多样本预测为正例,导致精确度较低。因此,F1得分作为精确度和召回率的综合指标,可以平衡这两个指标的表现。在评估模型性能时,通常会综合考虑精确度、召回率和F1得分。
Sigmoid函数
一种常用的激活函数,用于在神经网络中引入非线性。
它的数学表达式如下:
$$
sigmoid(x) = 1 / (1 + exp(-x))
$$
其中,$exp(-x)$表示e的-x次方,e是自然常数。
Sigmoid函数的输出值范围在0到1之间,通常用于将输入值映射到一个概率分布,或者作为二分类问题中的激活函数。
在GRU单元中,Sigmoid函数被用于计算两个门控向量:更新门(update gate)和重置门(reset gate)。这两个门控向量通过Sigmoid函数将输入向量和先前的隐藏状态向量映射到0到1之间的值,以控制它们对更新和重置操作的贡献。
更新门决定了先前的隐藏状态应该如何被保留或更新,而重置门决定了先前的隐藏状态如何与当前输入进行组合。
Sigmoid函数在GRU单元中通过限制门控向量的取值范围,使得GRU单元能够自适应地更新和遗忘信息,并有效地处理输入序列数据。
Hadamard乘积
也称为元素级乘积或逐元素乘积,是一种运算,用来对两个具有相同维度的向量、矩阵或张量进行逐元素的相乘。
对于两个维度相同的向量 A 和 B,Hadamard乘积的运算规则为:
$$
C = A ⊙ B
$$
其中 ⊙ 表示Hadamard乘积运算,C 是结果向量,C 的每个元素都等于 A 和 B 对应位置元素的乘积。
对于矩阵和张量,Hadamard乘积的运算规则与向量相同,只不过是在对应位置的元素进行相乘。
Hadamard乘积通常用于逐元素操作,如逐元素乘法、逐元素加法等。
它与矩阵乘法或点积运算不同,矩阵乘法是对应位置元素的乘积再求和,
而Hadamard乘积是对应位置元素直接相乘。
Hadamard乘积在深度学习中经常用于一些操作,如逐元素激活函数、逐元素损失函数、逐元素操作的正则化等。它可以帮助模型学习非线性关系,同时保持数据的维度不变。
Task2
前面了解了赛题,这个主要讲baseline代码,入门RNN和特征工程
解读官方baseline
set_random_seed
统一设置随机种子
1 | def set_random_seed(seed): |
这里做了这些操作
- 设置NumPy的随机种子
- 设置Python内置的随机数生成器的种子
- 设置PyTorch的随机种子
- 设置CUDA的随机种子
- 设置所有CUDA设备的随机种子
- 确保每次卷积算法选择是确定的
- 关闭CuDNN自动优化功能
就是把每一个自动优化或随机种子的选项都关掉了,然后确保结果不会因为自动优化或随机数而改变,因而可以复现结果。
SiRNADataset
1 | class SiRNADataset(Dataset): |
定义了一个SiRNADataset类来创建一个自定义的PyTorch数据集对象。
目的是将输入的数据框(df)中的序列数据分词、编码和填充,并返回编码后的序列和目标值。
SiRNADataset类的方法
初始化方法:
接受数据并处理成对象属性
接收了下面这些数据并保存为对象的属性:
1. 接收数据框(df)
2. 包含序列的列名(columns)
3. 词汇表(vocab)
4. 分词器(tokenizer)
5. 最大序列长度(max_len)
6. 否为测试集(is_test)
__len__方法
返回数据框中的样本数量。
__getitem__方法
根据给定的索引(idx),获取数据集中的第idx个样本。
首先根据索引获取数据框中的一行数据,然后对每一列的序列数据进行分词和编码。
- 如果是测试集模式(is_test为True),则返回编码后的序列。
- 如果不是测试集模式,则将目标值转换为张量,并返回编码后的序列和目标值。
tokenize_and_encode方法
接收一个序列(seq,这个就是我们要处理的序列)作为输入,根据序列是否包含空格,选择不同的方式分词。
这里有两种分词方法:
- 包含空格的序列,将其按空格进行分词; (这个就是对modified_siRNA_antisense_seq_list(modified_xxxx) 的数据,它本身已经根据空格分好了)
- 常规序列,使用指定的分词器进行分词。
然后,将分词后的token转换为词汇表中对应的索引,未知的token使用索引0(代表$
SiRNAModel 类
1 | class SiRNAModel(nn.Module): |
这个类继承自nn.Module类,用来处理RNA序列。
- 先将输入序列列表x传入嵌入层,
- 然后通过循环将每个序列的嵌入向量传入双向GRU层,取最后一个隐藏状态,并进行dropout处理。
- 最后,将所有序列的输出拼接起来,并传入一个全连接层,输出一个标量结果。
nn.Module类
nn.Module类是PyTorch中所有神经网络模型的基类,提供了一些基本的功能和方法,用于定义和管理神经网络模型的结构和参数。
nn.Module类的作用有:
定义模型的结构
通过__init__方法中定义各个层和模块,可以将不同的层组合在一起,构建出模型的结构。
前向传播函数
通过forward方法中定义前向传播的过程,可以将输入数据在模型中传递,计算输出结果。
参数管理
nn.Module类提供了一些方法,如parameters()和named_parameters(),可以自动追踪模型中所有的可学习参数,可以方便地进行参数的访问和管理。
parameters()
parameters()方法返回一个迭代器,该迭代器会遍历模型中的所有可学习参数。
可学习参数是指那些需要在训练过程中进行优化调整的参数,例如神经网络中的权重和偏置项。
1 | model = SiRNAModel(...) |
named_parameters()
named_parameters()方法返回一个迭代器,该迭代器会遍历模型中的所有可学习参数,并为每个参数附上一个名称。
这个方法在调试和模型分析时常见,可以方便地查看每个参数的名称和对应的数值。也可以利用这个方法来选择性地冻结或更新某些参数。
1 | for name, param in model.named_parameters(): |
模型保存和加载
nn.Module类提供了方法,如
state_dict()和load_state_dict(),可以方便地保存模型的状态和加载已保存的状态。
继承自nn.Module类的子类可以自由定义自己的网络结构,并且可以利用nn.Module提供的方法和功能来管理参数和实现前向传播过程。
还可以与优化器、损失函数、数据加载器等,进一步提升模型的训练和使用效果。
state_dict()
state_dict()(状态字典)是一个Python字典对象,其中包含了模型的所有可学习参数的名称和对应的张量值。
state_dict()方法返回模型的状态字典,可以将其保存到文件中,以便在之后的时间点恢复模型的状态。
1 | model = SiRNAModel(...) |
load_state_dict()
用于加载之前保存的模型的状态字典。可以将保存的状态字典加载到同一类别的模型对象中,以便恢复模型的参数。
1 | model = SiRNAModel(...) |
通过state_dict()和load_state_dict()方法,可以方便地保存和加载模型的参数状态,以便进行模型的训练和推理。这些方法在迁移学习、继续训练以及模型部署等场景中常见。
如何将序列转换成张量输入到模型里
关键代码是在
forward()方法
方法:forward(self, x)
参数: x (List[Tensor]): 输入序列列表
发现输入的是这个x,x又是输入的序列
1 | for inputs,target in train_loader: |
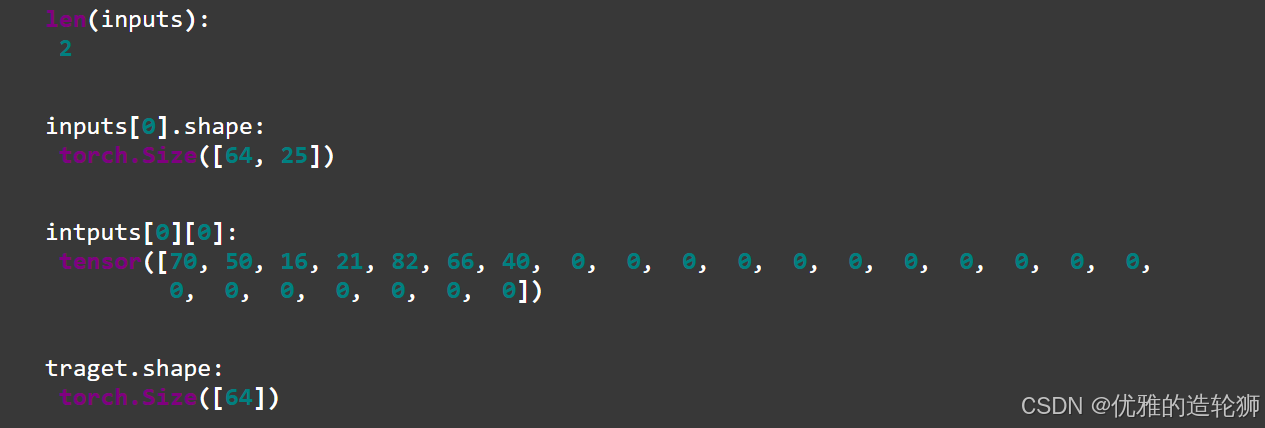
在处理siRNA序列数据时,我们首先注意到输入数据inputs包含两个元素,每个元素的尺寸为64×25。
这里的64表示批量处理的大小,而25代表每个序列的长度。通过观察inputs[0][0],我们可以了解到siRNA的反义链序列(siRNA_antisense_seq)在经过向量化处理后的表现。在这里,序列的前7位是非零值,这些非零值代表了序列编码中每个字符的唯一标识符。
在这个模型的嵌入层初始化时我们做了这样一个操作,
其中
vocab_size表示词汇表的大小
embed_dim表示嵌入向量的维度
padding_idx=0表示对应的填充符号的索引。
为了使RNN模型能够有效处理这些数据,需要保证每个输入样本的长度一致,在创建模型时采取了填充(padding)策略(上方引用)。
如果某个序列编码后的长度小于最大长度,我们会在其后补零,以确保所有序列在输入到RNN模型时具有统一的长度。
这里把所有序列都被填充至25位,来满足模型的输入要求。
如何为siRNA序列分配唯一标识符
首先进行分词处理
对于未格式化的siRNA_antisense_seq等序列
使用GenomicTokenizer实现
siRNA_antisense_seq序列通过每3个核苷酸一组划分,使用GenomicTokenizer实现,其中ngram和stride均设为3。
例如序列”AGCCGAGAU”,分词后得到[“AGC”, “CGA”, “GAU”]。
对于格式化的modified_siRNA_antisense_seq_list等序列
modified_siRNA_antisense_seq_list序列根据空格已分词。
基于数据集中所有token构建词汇表。
该词汇表映射token至唯一标识符,即索引。映射过程确保RNN模型接收数值形式输入,同时学习序列中不同token间关系。
使用GenomicVocab.create方法基于tokens创建基因词汇表。
创建基因词汇表代码
1 | # 创建分词器 |
- 先创建一个
GenomicTokenizer对象,用于对序列进行分词。 - 然后遍历数据集中的每个序列,如果序列中包含空格,则说明是修改过的序列,直接按空格切分并添加到
all_tokens中; - 如果序列中不包含空格,则使用分词器
tokenizer对序列进行分词,并将结果添加到all_tokens中。 - 最后使用
GenomicVocab.create方法基于all_tokens创建基因词汇表,设定最大词汇量为10000,词频阈值为1。
来获得序列的最大长度
1 | # 用于计算训练数据中每列数据最大长度 |
SiRNADataset类
完成上面的操作之后,在loader获取样本的时候把token转为索引,即我们通过转换成SiRNADataset类的过程中,让数据转换成索引
1 | class SiRNADataset(Dataset): |
这个类继承自PyTorch的Dataset类,用于加载数据并将其传递给模型进行训练或预测。
在__getitem__方法中,根据索引idx获取对应的数据行。然后针对每个包含序列的列,调用tokenize_and_encode方法对序列进行分词和编码。 如果是测试集模式,直接返回编码后的序列;如果是训练集模式,还需获取目标值并将其转换为张量。然后,tokenize_and_encode方法用于对序列进行分词和编码。对于常规序列,使用传入的分词器对其进行分词;对于修改过的序列,直接按空格进行分词。然后将分词后的token转换为词汇表中的索引,未知token使用索引0表示。最后将序列填充到最大长度,并返回张量格式的序列。
Dataset类
它是一个数据集的抽象接口,可以根据需要自定义数据集的读取和处理方式。
在使用PyTorch进行训练和预测时,需要将数据加载到Dataset对象中,并通过DataLoader对象对数据进行批处理和数据加载。
通过继承Dataset类,我们可以自定义数据集的处理逻辑,包括数据读取、数据预处理、数据转换等。
我们需要实现__len__和__getitem__方法,分别用于获取数据集的长度和获取指定索引位置的样本。
可以自定义Dataset类来灵活地处理不同类型的数据集,并将其传递给模型进行训练或预测。
关于训练的模型前面在SiRNAModel 类时讲过,就不再重述
我们首先进行索引嵌入处理,即将离散的符号(例如单词、字符或基因序列片段)转换成连续的向量形式。过程中涉及将高维的稀疏表示(如独热编码)转换为低维的密集向量,以使得语义相近的符号在向量空间中的相对位置更接近。
转换后,嵌入向量的维度将从BatchSize * Length扩展为BatchSize * Length * EmbeddingSize,其中EmbeddingSize,也就是嵌入维度embed_dim,被设定为200。
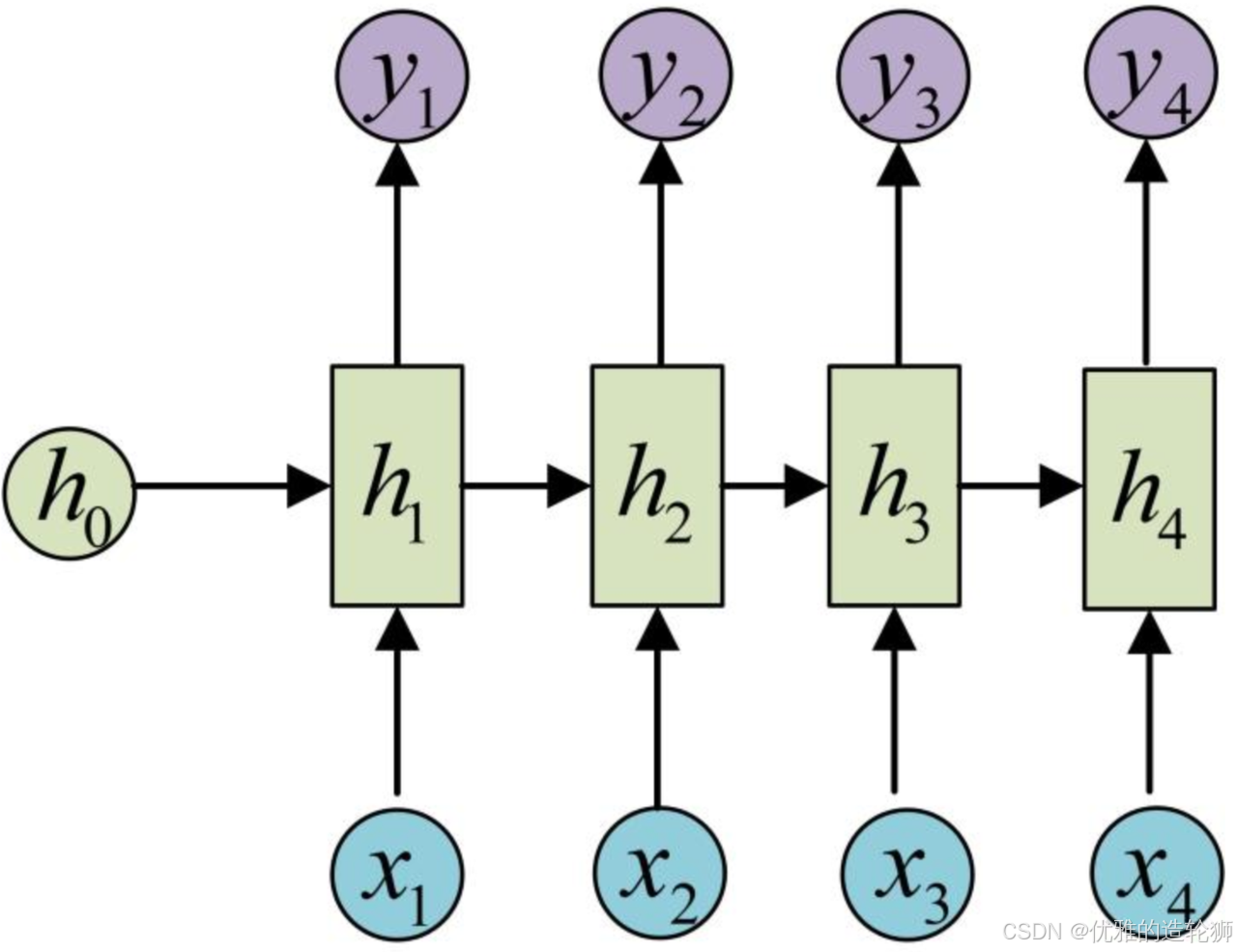
RNN(递归神经网络)知识点
一种专门用于处理序列数据的神经网络模型。
与传统的前馈神经网络不同,RNN具有反馈连接,可以将前面的输出作为后续输入的一部分,使其具有记忆性。
RNN的基本结构是一个单元(cell)或节点,其中包含一个输入层、一个隐藏层和一个输出层。隐藏层中的神经元通过时间反馈连接,使得信息可以在不同时间步之间传递和共享。这种结构使得RNN能够处理任意长度的序列数据,并且能够捕捉到序列中的上下文信息。
RNN的架构示意图
RNN,即循环神经网络(Recurrent Neural Network),是一种适合于序列数据的深度学习模型。它与传统的前馈神经网络(如多层感知机)不同,RNN 能够处理序列中的动态特征,即能够捕捉时间序列中的动态依赖关系。
RNN的数学表达可以简化为以下形式:
$$
h_t = f(W_{hh} h_{t-1} + W_{xh} x_t + b_h)
$$
$$
y_t = f(W_{hy} h_t + b_y)
$$
其中,
$h_t$是时间步$t$的隐藏状态。
$x_t$是时间步$t$的输入向量。
$W_{hh}$和$W_{xh}$分别是从上一个时间步的隐藏状态到当前隐藏状态、从当前时间步的输入到当前隐藏状态的权重矩阵。
$b_h$是隐藏层的偏置项。
$W_{hy}$是从隐藏状态到输出的权重矩阵。
$b_y$是输出层的偏置项。
$f$是激活函数。
RNN的训练过程
在RNN中,每个时间步都有一个输入和一个输出。输入可以是任意维度的向量,而输出通常是一个固定大小的向量或者是一个标量。RNN通过学习一组可学习的权重参数来对输入序列进行处理,并输出相应的预测结果。
RNN的训练过程通常是使用反向传播算法来优化模型的权重参数。 由于反向传播算法的梯度消失问题,在处理长序列时RNN往往会出现难以学习到长期依赖关系的情况。为了解决这个问题,一种常用的改进版本是长短期记忆网络(LSTM)和门控循环单元(GRU),它们能够更有效地捕捉和利用序列中的长期依赖关系。
RNN 的特点
- 循环连接:RNN的每个神经元不仅与下一层的神经元相连,而且与同一层的下一个时间步的神经元相连,形成了一个循环结构。
- 时间步:RNN在序列的每个时间步上都会进行计算,每个时间步的输出不仅依赖于当前的输入,还依赖于前一个时间步的输出。
- 隐藏状态:RNN通过隐藏状态(hidden state)来传递之前时间步的信息。隐藏状态可以看作是网络对之前序列信息的总结。
- 参数共享:在RNN中,同一网络参数在每个时间步上都会被重复使用,这简化了模型结构,但同时也带来了一些挑战,如梯度消失或梯度爆炸问题。
- 长短期记忆(LSTM)和门控循环单元(GRU):这两种网络结构是对传统RNN的改进,它们通过引入门控机制来解决梯度消失问题,使得网络能够学习长期依赖关系。
- 应用领域:RNN广泛应用于自然语言处理(NLP)、语音识别、时间序列预测等领域,特别是在需要处理序列数据和捕捉时间依赖性的任务中。
- 训练挑战:RNN的训练可能面临梯度消失或梯度爆炸的问题,这使得训练过程可能不稳定。现代优化技术如梯度裁剪或使用更高级的优化器(如Adam)可以帮助缓解这些问题。
- 变长序列处理:RNN能够处理不同长度的序列,但需要通过填充(padding)或截断来保证输入序列具有相同的长度。
数据的特征工程 (EDA)
在官方baseline中,得分较低可能是由于数据特征简单、序列特征构造粗糙以及数据量不足等原因。为了解决序列特征问题,可以将其转化为表格问题并进行特征工程。
基本操作
缺失值处理
检查数据是否存在缺失值,并根据具体情况决定如何处理缺失值,如删除、填充等。
异常值处理
检测和处理数据中的异常值,包括通过可视化和统计学方法识别异常值,并根据业务逻辑进行处理。
处理类别型变量
统计唯一值
1 | df.gene_target_symbol_name.nunique() |
计算DataFrame(df)中某一列(gene_target_symbol_name)中唯一值(unique value)的数量(nunique)。也就是统计该列中有多少不重复的值。
nunique()
nunique()函数是pandas库中的一个方法,用于计算一个序列(Series)或数据框(DataFrame)中唯一值的数量。
语法如下:
1 | Series.nunique(dropna=True) |
参数:
dropna:是否排除缺失值,默认为True,即排除缺失值。axis:对于数据框,可以指定按行(axis=0)或按列(axis=1)计算唯一值的数量。
1 | data = pd.Series([1, 2, 3, 2, 1, 4, 5, 2, 3]) |
统计每个值的频率分布
1 | df.gene_target_symbol_name.value_counts() |
这段代码是用来计算DataFrame(df)中某一列(gene_target_symbol_name)中每个唯一值(unique value)出现的次数(count)。它会返回一个Series对象,其中索引是唯一值,值是对应唯一值的出现次数。通过这个可以快速了解该列中每个值的频率分布。
value_counts()
value_counts()函数是pandas库中的一个方法,用于计算一个序列(Series)中每个唯一值的数量。
语法如下:
1 | Series.value_counts(normalize=False, sort=True, ascending=False, bins=None, dropna=True) |
参数:
normalize:是否返回相对频率,默认为False,即返回唯一值的数量。sort:是否按值进行排序,默认为True,即按值进行排序。ascending:是否按升序排列,默认为False,即按降序排列。bins:指定柱状图的箱数。dropna:是否排除缺失值,默认为True,即排除缺失值。
示例:
1 | data = pd.Series([1, 2, 3, 2, 1, 4, 5, 2, 3]) |
以上value_counts()方法计算了序列data中每个唯一值出现的次数,按降序排列输出。
one-hot特征的构造
1 | # 如果有40个类别,那么会产生40列,如果第i行属于第j个类别,那么第j列第i行就是1,否则为0 |
时间特征构造
有可能
没看出来,啃臭cv一份,很妙
在数据观察的时候发现,siRNA_duplex_id的编码方式很有意思,其格式为AD-1810676.1,我们猜测AD是某个类别,后面的.1是版本,当中的可能是按照一定顺序的序列号,因此可以构造如下特征
1 | siRNA_duplex_id_values = df.siRNA_duplex_id.str.split("-|\.").str[1].astype("int") |
这段代码是从siRNA_duplex_id列中提取出按照一定顺序的序列号作为新的特征siRNA_duplex_id_values。
siRNA_duplex_id的编码格式为”AD-1810676.1”,其中”AD”表示某个类别,”.1”表示版本号,而中间的数字则是按照顺序的序列号。(假定,大概率)
代码通过使用正则表达式分隔符”-“和”.”,将siRNA_duplex_id拆分成多个部分,然后取第二部分(索引为1),并将其转换为整数类型。得到的siRNA_duplex_id_values列即为按照一定顺序的序列号特征。
上述对每一个siRNA_duplex_id的过程同下
(方便复制)
1 | import re |

包含某些单词
对df中的cell_line_donor列构造特征
1 |
|
代码小结
- 使用
pd.get_dummies()函数对cell_line_donor列进行独热编码, 编码后的列会根据不同的取值创建新的列。 - 使用列表推导式为
df_cell_line_donor的列名添加前缀 “feat_cell_line_donor_”。 - 创建新的特征列
feat_cell_line_donor_hepatocytes,根据cell_line_donor列是否包含 “Hepatocytes” ,将布尔值转换为整数(1 表示包含,0 表示不包含)。 - 创建新的特征列
feat_cell_line_donor_cells,根据cell_line_donor列是否包含 “Cells” 来确定的,将布尔值转换为整数(1 表示包含,0 表示不包含)。
将 cell_line_donor 列转换为独热编码,并创建两个新的特征列,用于表示是否包含特定的关键词。
对碱基的模式进行特征构造
根据上一个task中的rna知识提取
1 | def siRNA_feat_builder(s: pd.Series, anti: bool = False): |
代码小结
- “feat_siRNA_{name}_seq_len”:siRNA序列的长度作为特征。
- siRNA序列的第一个和最后一个位置,在前端或后端:
- “feat_siRNA_{name}seq{c}_{‘front’ if pos == 0 else ‘back’}”:判断序列的第一个或最后一个碱基是否与’A’, ‘U’, ‘G’, ‘C’相等。
- siRNA序列的起始和结束:
- “feat_siRNA_{name}seq_pattern_1”,…,”feat_siRNA{name}_seq_pattern_8”:判断序列是否以特定的模式开头和结尾。
- siRNA序列的第二位和倒数第二位:
- “feat_siRNA_{name}_seq_pattern_9”:判断序列的第二位是否为’A’。
- “feat_siRNA_{name}_seq_pattern_10”:判断序列的倒数第二位是否为’A’。
- “feat_siRNA_{name}_seq_pattern_GC_frac”:计算序列中的GC碱基占整体长度的比例。
最后选择模型预测
这里是task2给出的lightgbm的代码来对特征值预测 引一份
1 | train_data = lgb.Dataset(X_train, label=y_train) # 创建训练数据集,X_train为特征矩阵,y_train为标签向量 |
分数
一些常用的LightGBM参数
LightGBM是一种梯度提升树模型。
boosting_type
指定梯度提升树的类型
有gbdt(传统的梯度提升决策树)、dart(dropout加速梯度提升树)和goss(梯度优化送出采样)。
objective
指定模型的优化目标,根据任务类型选择合适的目标函数。
回归任务可以使用regression,分类任务可以使用binary或multiclass。
metric
指定模型的评估指标,用于衡量模型的性能。
对于回归任务可以使用root_mean_squared_error(均方根误差)。
max_depth
每棵树的最大深度,控制模型的复杂度。
较小的值可以防止过拟合,但可能会导致欠拟合。
max_depth 一般在 (6,10)
learning_rate
学习率控制每个树的贡献。
较小的值会使算法收敛得更慢,但可能会获得更好的精度。
num_boost_round
迭代次数,指定生成的树的数量。
较大的值可以提高模型的性能,但也会增加计算时间。
valid_sets
用于验证模型的数据集,可以根据需要指定多个。
在训练过程中,模型会根据验证集的性能进行调整。
callbacks
在训练过程中执行的回调函数,可以用于打印模型的验证结果、保存模型等。
可以通过回调函数自定义返回的东西,如打印测试情况之类的
这里就是回调时,用了print_validation_result 作为输出
输出函数
2
3
result = env.evaluation_result_list[-1]
print(f"[{env.iteration}] {result[1]}'s {result[0]}: {result[2]}") # 打印验证结果的回调函数,用于输出每次迭代后的验证结果
num_leaves
每棵树的叶子节点数,与
max_depth参数一起控制模型的复杂度。
min_data_in_leaf
叶子节点的最小数据量,用于防止模型在小数据集上过拟合。
subsample
训练时使用的样本比例,可以用于防止过拟合。
verbose
是否在训练过程中打印详细的信息。
random_state
随机数生成器的种子,用于确保结果的可复现性。
device_type
指定训练时使用的设备类型,如CPU或GPU。
一般本地训练需要调整
更多参数还是建议自主参考官方文档
Parameters — LightGBM 4.5.0.99 documentation
https://lightgbm.readthedocs.io/en/latest/Parameters.htmll
举例一个其他的模型的训练参数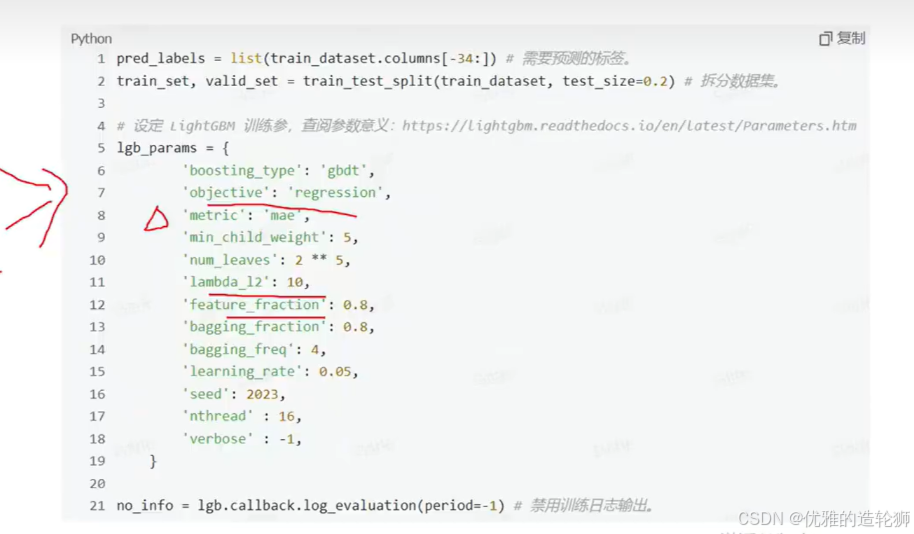
根据具体任务和数据的特点,可以尝试不同的参数组合来优化模型性能。
tips : 可以构造更多特征,多模型融合,k折 ,调超参等方法涨点
这里给出一个k折的框架
1 | from sklearn.model_selection import KFold |
分数
目前还在冲分,后续补上代码

Task3
特征工程进阶
对task2引入生物知识
引入的长度、GC含量等特征细节刻画
1 | def siRNA_feat_builder3(s: pd.Series, anti: bool = False): |
代码可以看出,新增有长度分组,GC含量和局部GC含量
修饰siRNA构建特征
1 | def siRNA_feat_builder3_mod(s: pd.Series, anti: bool = False): |
修饰siRNA序列进行n-gram的词频统计
同时也可对未修饰序列进行相同的操作
1 | class GenomicTokenizer: |
1 | # 处理序列特征 |
tokenizer 的工作方式
这里解释一下通过一个例子来展示不同 tokenizer 的工作方式。
我们有一个由碱基组成的基因序列:
1 | AGTCATG |
我们将使用这个序列来演示每个 tokenizer 如何将其分割。
tokenizer1 (ngram=1, stride=1)
- 将序列分割成单个碱基的片段,步长为1。
- 结果:
['A', 'G', 'T', 'C', 'A', 'T', 'G']
tokenizer2 (ngram=2, stride=2)
- 将序列分割成长度为2的片段,步长为2。
- 结果:
['AG', 'TG'](从’A’开始,跳过一个碱基到’G’,然后再次跳过一个碱基到’T’)
tokenizer3 (ngram=3, stride=3)
- 将序列分割成长度为3的片段,步长为3。
- 结果:
['AGT'](从’A’开始,跳过两个碱基到’G’)
tokenizer6 (ngram=6, stride=6)
- 由于序列长度只有7个碱基,而步长为6,所以这个
tokenizer只会生成一个长度为6的片段。 - 结果:
['AGTCAT']
1 | # 子串词频统计,修饰序列 |
siRNA序列与target序列对比
1 | from Bio import pairwise2 |
如果siRNA是antisense类型
result_df = get_feat_align(df, anti=True)
如果siRNA是sense类型
result_df = get_feat_align(df)
其他生物特征
有重复
lgb模型优化
低Remaining范围样本高权重
1 | weight_ls = np.array(feats['mRNA_remaining_pct'].apply(lambda x:2 if ((x<=30)and(x>=0)) else 1)) |
这段代码是将feats中的mRNA_remaining_pct列的值进行一些判断和处理,生成一个新的weight_ls数组。
这段代码根据mRNA_remaining_pct列的值是否在0到30之间,将对应位置上的weight_ls值设置为2或者1。
使用官方评价指标作为损失函数
由原来的root_mean_squared_error评价指标被替换为更加复杂的官方评价分数
具体公式为:
$$\text{score} = 50% \times \left(1 - \frac{\text{MAE}}{100}\right) + 50% \times F1 \times \left(1 - \frac{\text{Range-MAE}}{100}\right)$$
1 | # calculate_metrics函数用于计算评估指标 |
自适应学习率
1 | # adaptive_learning_rate函数用于自适应学习率 |
多折交叉训练
1 | # train函数用于训练模型 |
完成以上操作的分数
超参数优化
贝叶斯优化(推荐)
您可以使用如optuna这样的库来执行贝叶斯优化超参数, 参考代码如下
伪代码:
1 | import optuna |
通过贝叶斯优化方法来进行超参数优化。
- 拆分数据集为训练集和测试集。
- 定义超参数搜索空间。
- 创建评估函数,训练LGBMClassifier模型,计算准确率。
- 使用贝叶斯优化,找出最佳超参数。
真的跑很久。。。还没跑完
网格搜索(Grid Search)
使用LightGBM
伪代码:
1 | from sklearn.model_selection import GridSearchCV |
随机搜索(Random Search)
使用LightGBM
伪代码:
1 | from sklearn.model_selection import RandomizedSearchCV |
集成学习
多模型可以结合使得稳定
集成学习就是把多个弱分类器或回归模型组合起来,变成一个强分类器或回归模型,从而提高预测的准确性。
实现集成学习的方式有很多种,比如通过投票决定最终结果、取平均值来预测、或者给每个模型分配不同的权重。集成学习的主要思想是通过多个模型之间的合作,来弥补每个模型的不足,使整体模型的预测能力更强。
常见的集成学习方法:
- Bagging(自助聚合):通过在原始数据集上进行多次重采样来创建多个子集,分别训练多个模型,最后进行平均或多数投票决策。
- Boosting:训练多个模型,每个模型都尝试纠正前一个模型的错误,通常是序列处理。
- Stacking:训练多个不同的模型,然后再训练一个新的模型来综合这些模型的输出。
Stacking
Stacking 是一种集成学习技术,它将多个模型的预测结果作为输入,然后使用另一个模型(通常称为元模型或元分类器)来进行最终的预测。
举例一个使用Python的 scikit-learn 库实现Stacking:
伪代码:
1 | from sklearn.datasets import load_iris |
Stacking模型是什么?
想象一下,你有好几个不同的老师,他们每个人都对同一组学生进行考试评分。Stacking模型就像是一个“超级老师”,它收集这些不同老师给的分数,然后根据这些分数再给出一个最终的评分。
基模型
在这个例子里,我们有三个“老师”:
- 第一个老师用的是“随机森林”方法来评分。
- 第二个老师用的是“梯度提升”方法。
- 第三个老师用的是“支持向量机”方法。
元模型
然后,我们有一个“超级老师”,也就是我们的元模型,它用的是“逻辑回归”方法来根据前面三个老师的评分给出最终的评分。
为什么要这么做?
- 有时候,不同的老师(模型)对同一组学生(数据)的看法会有所不同。通过综合他们的意见,我们可以得到一个更全面、更准确的评分。
- 但是,这也有风险,如果这些老师(模型)都倾向于犯同样的错误,那么“超级老师”也可能跟着犯错。
如何实现?
- 我们首先把学生(数据)分成两部分:一部分用来让每个老师单独评分(训练集),另一部分用来测试最终的评分结果(测试集)。
- 每个老师都用他们的方法给训练集的学生打分。
- 然后,我们把这些分数收集起来,让“超级老师”来根据这些分数给出最终的评分。
- 我们用测试集来看看“超级老师”的评分有多准确。
注意事项
- 支持向量机老师需要一个特别的设置(
probability=True),这样它才能给出每个学生可能得到每个分数的概率,这对于“超级老师”来说很重要。 - 我们要小心,不要让“超级老师”太复杂,否则它可能会过度拟合,也就是说,它可能只是在模仿训练集中的分数,而不是真正理解学生的能力。
总的来说,Stacking模型是一种很有趣的方法,可以让我们把不同的模型结合起来,得到更好的预测结果。但是,我们也需要小心,确保它不会变得太复杂,导致在新数据上表现不佳。
官方给出的lgb举例
假设已有LightGBM、XGBoost和一个简单的神经网络模型,下面是一个使用Stacking方法的Python示例代码:
1 | import numpy as np |
混合学习
在解决复杂的生物信息学问题时,机器学习和深度学习的混合方法可以提供强大的工具。
这种方法包括两个主要部分:使用深度学习模型进行特征提取,然后使用传统的机器学习模型进行最终的决策。可以使用PyTorch构建深度学习部分,然后将输出特征传递给LightGBM进行分类或回归。这种混合方法结合了深度学习的特征学习能力和传统机器学习模型的效率与解释性,可以在生物信息学问题中提供强大的解决方案。
构建PyTorch模型
首先,我们定义一个简单的卷积神经网络(CNN)来处理序列数据。这个模型将用于提取有用的特征。
1 | import torch |
使用LightGBM进行回归
在获取特征后,我们可以使用LightGBM进行回归预测。
1 |
|
其他思路
- 集成额外的生物信息学数据库来增强特征。
- 实施自动化特征选择流程以减少模型复杂性和过拟合。
- 尝试动态调整学习率,如学习率预热和循环学习率。
- 考虑多目标优化,同时优化不同评价指标或设计更全面的评价函数。
- 生物学角度新特征
新模型构建
- 使用attention机制进行end2end的建模,将siRNA序列和target gene序列进行拼接,并捕捉它们之间的相关模式。
- 利用现有的生物序列基础模型,生成siRNA和target gene序列的表征向量,并将其输入模型以提高预测效果。
外部数据集(官方未禁用)
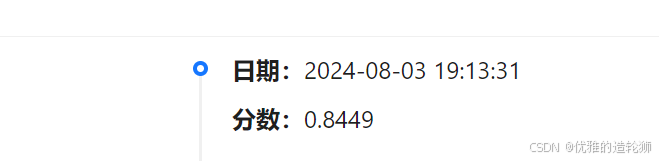
笔记发布前最新成绩
引用文档
siRNA和shRNA:通过基因沉默抑制蛋白表达的工具
http://www.labome.cn/method/siRNAs-and-shRNAs-Tools-for-Protein-Knockdown-by-Gene-Silencing.html
Datawhale
https://linklearner.com/activity/12/4/16
Datawhale
https://linklearner.com/activity/12/4/11
Datawhale
https://linklearner.com/activity/12/4/5
Datawhale
https://linklearner.com/activity/12/4/4